유전자치료의 새로운 패러다임, 세포소기관 염기교정 기술
조성익 교수 (KAIST)
1. 개요
염기교정(Base editing)은 DNA 서열을 가위로 자르지 않고 특정 염기를 원하는 다른 염기로 치환함으로써, 문서의 오탈자를 고치듯 유전 정보를 정밀하게 수정하거나 새 정보를 삽입할 수 있는 기술이다. 질환을 유발하는 돌연변이 염기를 정상 염기로 복구할 수 있어, 유전질환 치료·암 연구·농업 형질 개량 등 여러 분야에서 패러다임 전환을 이끌고 있다.
유전자교정 시장은 연평균 15% 이상 성장하고 있으며(그림 1), 특히 정밀의료와 농업 응용 부문에서 실용화 가능성이 높다. 유전자교정 분야를 선도하고 있는 CRISPR 시스템은 gRNA만 갈아 끼우면 원하는 유전자를 자유자재로 표적할 수 있어, 뉴클레이스·염기교정기·프라임교정기까지 줄줄이 개발되며 유전자교정 기술을 빠르게 진화시켜 왔다. 이론적으로는 거의 모든 염기를 마음대로 고칠 수 있는 시대가 열린 셈이다.
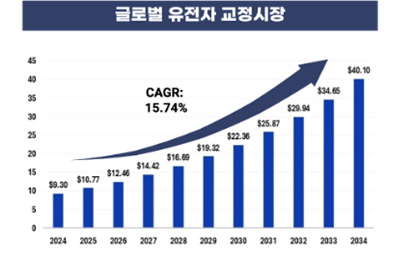
그림 1. 글로벌 유전자교정 시장 성장
출처: Precedence Research
하지만 이 강력한 도구로도 아직 손이 닿지 않는 영역이 남아 있다. 대표적인 예가 미토콘드리아와 엽록체 같은 세포소기관 유전체다.
미토콘드리아는 세포공생설의 산물로, 고대 원핵생물이 진핵세포 속으로 들어와 공존하게 된 결과물이다. 오늘날에는 우리 몸에서 ATP를 대량으로 만들어 내는 ‘에너지 공장’으로 자리 잡아, 모든 세포의 에너지를 담당하고 있다.
이 소기관은 자체 유전체인 미토콘드리아 DNA(mtDNA)를 보유하는데, mtDNA는 이중막 구조로 둘러싸인 미토콘드리아 매트릭스 안에 위치하고 있다. 미토콘드리아의 기능은 핵 DNA와 mtDNA 양쪽에서 조절되는 복잡한 유전적 상호작용에 의해 결정된다.
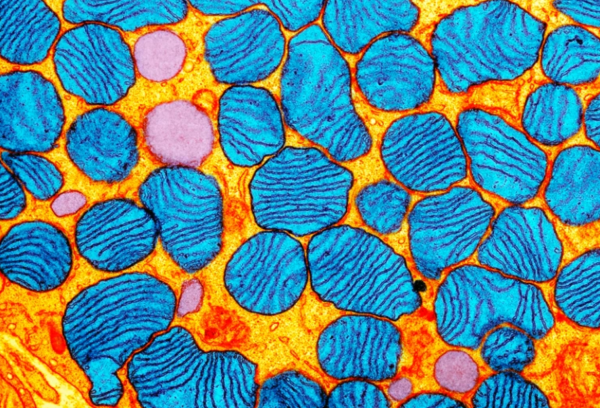
인간 mtDNA는 16,569bp의 이중가닥 원형 DNA로, 산화적 인산화(OXPHOS)에 필수적인 37개의 유전자를 담고 있다. 여기에는 13개의 OXPHOS 단백질 복합체 소단위, 2개의 리보솜 RNA, 22개의 전사 RNA가 포함되어 있으며 미토콘드리아 번역에 관여하게 된다. 핵 DNA가 세포당 두 사본을 갖는 것과 달리, mtDNA 사본은 세포 유형에 따라 다양하며 최대 10만 사본까지 존재할 수 있다(그림 2).
그림 2. 미토콘드리아 세포소기관 TEM 사진
출처: Nature (2020)
그러나 문제는 이 작은 에너지 공장의 유전체에 변이가 일어나면 시각 상실(레버씨 시신경병증, LHON)이나 뇌 변성(Leigh 증후군)과 같은 중증 질환이 발생한다는 점이다. 이러한 미토콘드리아 질환 변이의 90% 이상이 단일 염기 변화이고, 신기하게도 그중 85%가 C→T 또는 A→G여서, 염기교정기가 소기관 내부로 들어가기만 하면 변이 교정과 모델링이 상당수 가능해진다.(그림 3).
그림 3. 미토콘드리아 유전질환의 Genotype
그러나 결정적인 걸림돌이 있다. 단백질은 미토콘드리아 막을 통과할 수 있어도, CRISPR 시스템의 핵심 열쇠인 gRNA가 소기관 안으로 이동한 사례는 아직 없다. gRNA가 이동하지 못하면 CRISPR 계열 유전자교정 기술은 제대로 작동할 수 없다. 이 때문에 기존 CRISPR 기반 시도들은 번번이 실패했고, 미토콘드리아 유전자교정은 여전히 미개척지로 남아 있다. 결국 소기관 유전체를 표적, 활용할 수 있는 새로운 교정 전략이 요구되는 상황이다.
본 글에서는 CRISPR의 한계를 넘어서는 세포소기관 염기교정 기술을 간략히 소개하고자 한다.
2. 최초의 세포소기관 염기교정.. 시토신 염기교정기 ‘DdCBE’
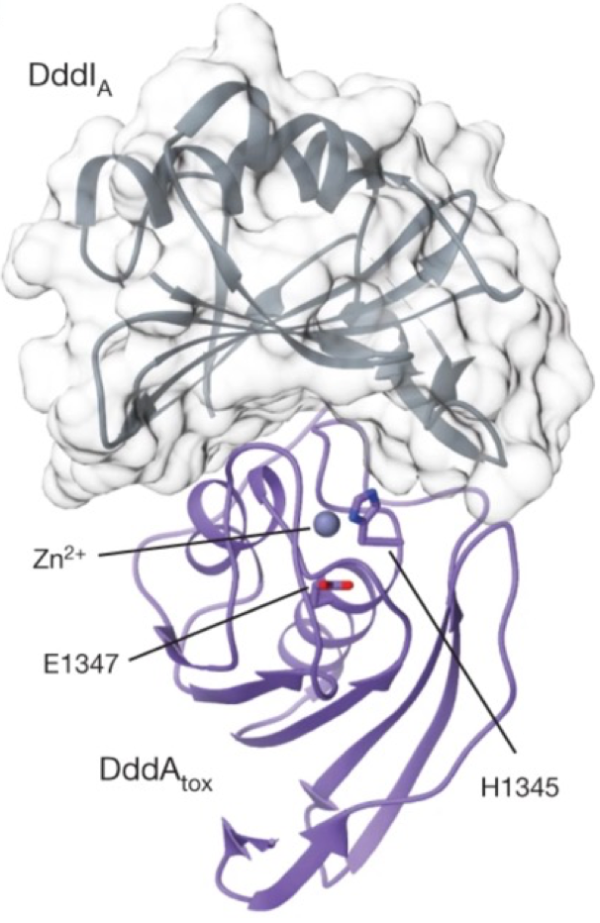
세포소기관 유전자교정 기술의 첫 주자는 시토신 염기교정기다. 그 시작점은 바로 세균 독소 DddAtox의 발견이다(그림 4). 기존에 보고된 탈아민효소는 모두 단일가닥 DNA에서만 작동했기 때문에, CRISPR 시스템과 결합해 gRNA가 노출한 단일가닥을 교정하는 방식으로만 활용됐다. 그러나 gRNA가 들어갈 수 없는 미토콘드리아, 엽록체 같은 세포소기관에서는 TALE이나 징크핑거 단백질만으로 이중가닥 DNA를 풀어낼 수 없어, 기존 효소를 융합해도 교정이 불가능했다. 이러한 한계를 DddAtox가 돌파한 것이다.
그림 4. 최초 DNA 이중가닥 시토신 탈아민효소
출처: Nature (2020)
DddAtox는 이중가닥 DNA에서도 C를 U로 탈아민화할 수 있는 최초의 효소로, 이를 바탕으로 세포소기관 염기교정을 가능하게 만들었다. 다만 독성이 강해 그대로는 사용할 수 없어, 연구진은 DddAtox를 두 조각으로 나눠(split) 각각을 무해·비활성 상태로 만든 뒤, 서로 인접한 표적 염기에 결합하도록 맞춤형 TALE 단백질에 부착했다. 여기에 변환된 U가 제거되지 않도록 uracil glycosylase inhibitor(UGI)를 더해 RNA가 필요 없는 시토신 염기교정기 ‘DdCBE’를 완성했다(그림 5).
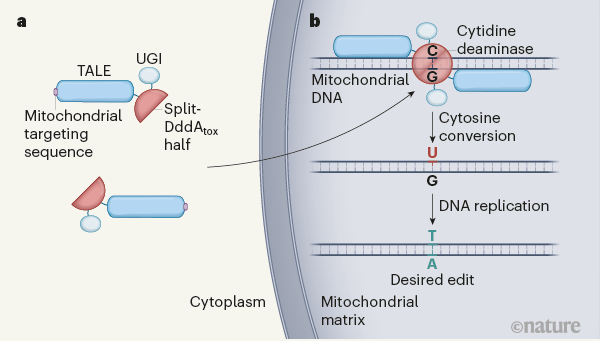
그림 5. 세포소기관 시토신 염기교정기 DdCBE
출처:Nature (2020)
이 시스템은 gRNA 없이도 인간 미토콘드리아 DNA에서 C·G를 T·A로 정확히 바꾸었고, 질환 관련 변이를 세포 모델에 재현해 산소 소비 감소와 OXPHOS 기능 변화를 확인할 수 있었다. 결국 DddAtox의 발견과 분할·재조합 전략은 미토콘드리아 유전체 교정을 가능하게 한 첫 시토신 염기교정기의 탄생으로 이어졌다.
3. DdCBE의 한계를 넘어..아데닌 염기교정기 ‘TALED’
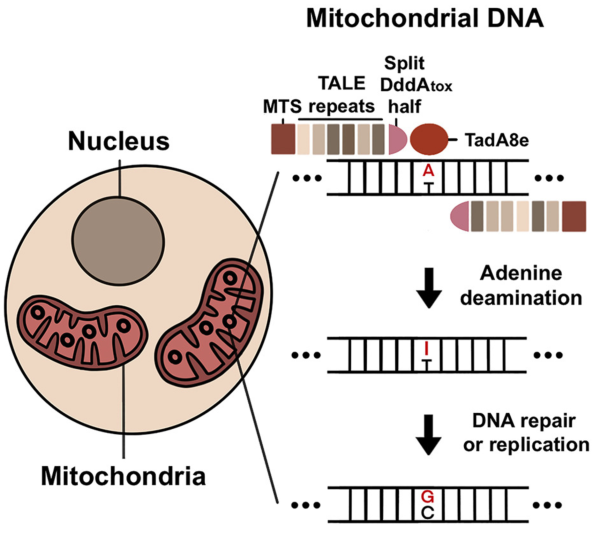
시토신 염기교정 기술의 한계를 극복한 것은 바로 아데닌 염기교정기 ‘TALED’다. 미토콘드리아 돌연변이의 약 90% 중 절반은 시토신 염기교정기로 해결할 수 있지만, 나머지 절반은 반드시 아데닌 염기교정이 필요하다. 게다가 1세대 기술인 DdCBE는 5′-TC 서열에서만 C→T 변환이 가능해 전체 mtDNA 변이의 8분의 1만 겨냥할 수 있었다. TALED는 이 한계를 정면으로 돌파했다.
아데닌 탈아민효소 TadA는 단일가닥 DNA에서만 작동하므로, 이중가닥 mtDNA를 열어 줄 도우미가 필요하다. TALED는 Burkholderia cenocepacia 유래 DddA를 촉매력이 낮은 형태로 변이시키거나 두 조각으로 분할해 독성을 제거한 뒤, 맞춤형 TALE 배열로 표적 서열에 정밀 도킹시킨다. 이어 엔지니어드 TadA를 결합해 노출된 아데닌을 I(결국 G)로 바꾼다. 이 설계에서는 DddA가 본래 시토신 탈아민효소임에도 이중가닥 DNA를 열어 주는 역할만 하고, uracil glycosylase inhibitor(UGI)를 넣지 않아 C→T 반응이 일어나지 않으므로 오로지 A→G만 교정된다(그림 6).
그림 6. 미토콘드리아 아데닌 염기교정기 TALED
출처:Cell (2022)
gRNA가 불필요하므로 미토콘드리아 내 RNA 수송 문제를 피할 수 있고, TALE 특이성 덕에 원하는 위치만 정밀하게 표적할 수 있다. 실제로 사람 세포에서 맞춤형 TALED는 표적 A를 G로 고효율 교정해 질환 관련 변이를 정확히 재현했으며, 이는 mtDNA 변이 연구와 미토콘드리아 유전질환 치료 전략에 새로운 장을 열었다.
4. 3차 구조 기반 엔지니어링을 통한 고정밀 TALED
2022년, 세포주에서 세포 사멸이 일어나고, 생쥐 배아에서 blastocyst 형성이 실패(arrest)하는 등, TALED 기술의 독성이 최초 보고됐다. 하지만 흥미롭게도 기존 세포소기관 시토신 염기교정기인 DdCBE와 TALED는 DNA 결합 단백질(TALE)과 시토신·아데닌 디아미네이스 연결 방식까지 거의 동일한 구조를 갖지만, DdCBE에서는 이러한 배아 독성이 전혀 나타나지 않았다.
연구진은 이 현상이 두 기술 간의 유일한 차이점, 즉 TALED에만 포함된 아데닌 탈아민효소 TadA8e의 특이적 활성에 기인한다고 판단하였다. 이에 독성의 근본 원인을 규명하기 위해 TadA8e의 작용 특성을 면밀히 분석하고, 이를 저독성·고정밀 변이체로 재설계하는 후속 연구를 시작하게 되었다.
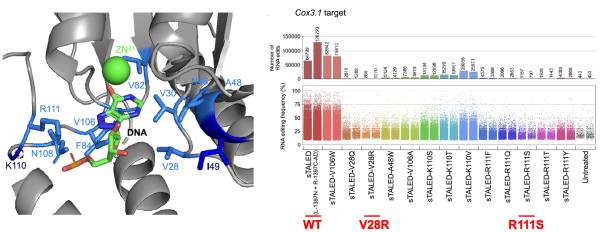
실제로 야생형 TALED(WT TALED)를 HEK293T 세포에 발현하자 전사체 전역에서 약 10만 개의 A→I 오프타겟(off-target)이 검출되어 CRISPR계 ABE8e(약 4000개)보다 수십 배 높은 RNA 오프타겟 활성을 나타냈다. 이에 3차 구조 정보를 활용해 TadA8e 기질 결합 포켓의 11개 아미노산을 모두 치환한 209종 라이브러리를 제작 및 스크리닝했고 그 결과, RNA 오프타겟 수를 96,559개에서 852개 및 829개로 약 99% 감소시키면서도 DNA 온타겟 효율은 유지하는 TAELD-V28R과 TALED-R111S 변이체를 선별할 수 있었다(그림 7).
그림 7. TALED의 효소 활성 부위 3차 구조 (왼쪽),
V28R과 R111S 변이체를 포함하는 TALED 변이체의 RNA off-target (오른쪽)
출처:Cell (2024)
5. TALED 변이체 활용, 세계 최초 미토콘드리아 질환 모델
이후 고성능 TALED 변이체(V28R·R111S)를 활용한 미토콘드리아 질환 모델이 제작됐다.
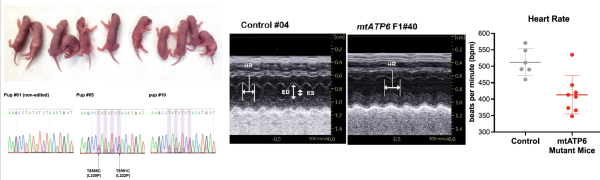
WT TALED를 투입한 배아는 염기교정 후 모두 arrest되어 blastocyst에 도달하지못 했으나, Leigh 증후군 연관 m.T8591C 병원성 변이를 정확히 삽입한 배아는 모두 정상적으로 blastocyst로 분화했다. 또한 교정 범위(window)가 좁아짐으로써 불필요한 bystander 변이도 크게 줄어 단일 염기교정이 우세하게 일어났다.
교정된 마우스는 산소 소비율과 ATP 생성이 감소하고 심박수가 느려지는 등 Leigh-유사 대사·심장 이상이 확인되었다. TadA8e의 기질 결합 부위를 정밀 설계해 TALED 독성 문제를 해소한 생체 질환 모델 제작까지 성공한 것이다.
그림 8. TALED 변이체로 교정된 배아에서 태어난 질환 마우스(왼쪽),
질환 마우스에서의 심장 초음파 사진(중간)과 느려진 심장 박동수(오른쪽)
출처:Cell (2024)
6. CRISPR계 염기 교정에도 적용 가능한 TadA 변이체
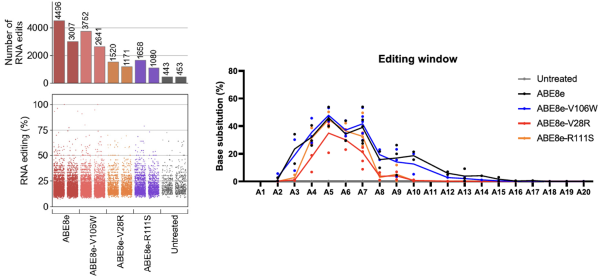
미토콘드리아 질환 마우스 모델에서 확인한 TALED의 교정 효율을 CRISPR계 교정기에 활용할 수 있을지 알아보기 위해, TadA 변이체인 TadA8e-V28R과 TadA8e-R111S를 CRISPR-Cas9 기반 아데닌 염기교정기에 적용해 보았다. 그 결과, 표적 위치에서의 A→G 교정 효율은 기존 ABE8e와 거의 동등하게 유지되었다. 동시에 불필요한 DNA bystander 변이가 크게 줄었으며, 전사체 RNA에서 A→I 오프타겟 또한 크게 감소했다.
이로써, TadA 변이체가 미토콘드리아 ABE 뿐 아니라, 핵에서 널리 사용되고 있는 CRISPR계 ABE에서도 정밀한 염기교정이 가능함을 확인하였다.
그림 9. TadA8e-V28R과 TadA8e-R111S가 적용된 CRISPR계 ABE의 전사체 off-target(왼쪽) 및 교정 window(오른쪽)
출처:Cell (2024)
7. 맺음
CRISPR 기술의 비약적 발전으로 더이상 교정하지 못할 유전자가 없을 듯 보였지만, gRNA를 세포소기관으로 전달하기 어렵다는 제약 탓에 세포소기관 유전체 교정은 여전히 미개척 영역으로 남아 있었다.
DdCBE와 TALED의 등장으로 미토콘드리아 단일염기 변이를 직접 교정할 수 있는 길이 열렸지만, 이는 세포소기관 염기교정 기술 개발의 시작에 불과하다.
현재 유전자교정 기술 엔지니어링, 서열 범위 축소, 오프타겟 최소화, 단백질 및 mRNA 전달 플랫폼 고도화 같은 연구가 활발히 진행되고 있다. 이러한 움직임은 미토콘드리아를 넘어 엽록체에도 적용돼 광합성 효율 개선 및 작물 형질 개량으로 이어지고 있다.
결국 세포소기관 염기교정은 희귀 미토콘드리아 질환 치료제와 고성능 농업 소재 개발 모두를 현실화할 핵심 기술로 자리매김하고 있으며, 미래 시장 및 임상·산업적 활용 가능성은 더욱 확대될 것으로 보인다.
8. 참고문헌
[1] Russell, O. M., et al. (2020). "Mitochondrial Diseases: Hope for the Future." Cell 181(1): 168-188.
[2] Mok, B.Y., de Moraes, M.H., Zeng, J.et al.A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing. Nature 583, 631–637 (2020). https://doi.org/10.1038/s41586-020-2477-4
[3] Lim, K. (2024). "Mitochondrial genome editing: strategies, challenges, and applications." BMB Rep 57(1): 19-29.
[4] Sung-Ik Cho*, Kayeong Lim*, Seongho Hong*, Jaesuk Lee, Annie Kim, Ji Min Lee, Young Geun Mok, Eugene Chung, Seunghun Han, Sang-Mi Cho, Jieun Kim, Sanghun Kim, Eun-Kyoung Kim, Ki-Hoan Nam, Yeji Oh, Minkyung Choi, Seonghyun Lee#, Hyunji Lee#, Jin-Soo Kim# (2024) Engineering TALE-linked deaminases to facilitate precision adenine base editing in mitochondrial DNA. Cell
[5] Sung-Ik Cho, Seonghyun Lee, Young Geun Mok, Kayeong Lim, Jaesuk Lee, Ji Min Lee, Eugene Chung and Jin-Soo Kim (2022) Targeted A-to-G base editing in human mitochondrial DNA with programmable deaminases. Cell
[6] Liang Chen, Changming Luan, Mengjia Hong, Meng Yuan, Hao Huang, Debo Gao, Xinyuan Guo, Zhengxin Chen, Yongmei Li, Lei Yang, Zongyi Yi, Wensheng Wei, Mingyao Liu, Liangcai Gao, Honghui Han, Dali Li. A mitochondrial disease model is generated and corrected using engineered base editors in rat zygotes.Nat Biotechnol 2025
[7] Zhang, X., Zhang, X., Ren, J.et al.Precise modelling of mitochondrial diseases using optimized mitoBEs. Nature 639, 735–745 (2025). https://doi.org/10.1038/s41586-024-08469-8
[8] Chen, L., Hong, M., Luan, C.et al. Efficient mitochondrial A-to-G base editors for the generation of mitochondrial disease models.Nat Biotechnol(2025). https://doi.org/10.1038/s41587-025-02685-x
* 본 기고문은 과학기술정보통신부에서 시행한 「유전자편집·제어·복원기반기술개발사업」수행의 일환으로 작성되었습니다.
* 본 내용은 유전체편집연구지원사무국(GERC)의 의견과 다를 수 있음을 밝힙니다.
* 인용, 발표하실 때에는 반드시 출처를 밝혀주시기 바랍니다.